анных
29 июня 2017 г.
Кратко
В этом письме к добровольцам д-р Алессандра Карбоун, главный исследователь проекта Help Cure Muscular Dystrophy ("помогите вылечить мышечную дистрофию") объявляет о выпуске двух новых инструментов в открытом доступе, которые могут ускорить работу исследователей, работающих над взаимодействием белков.
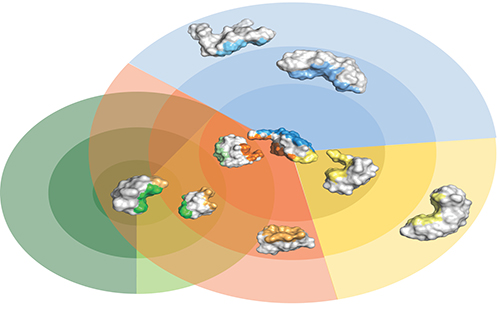
Картинка выше - рендеринг белковой структуры с тремя участками взаимодействия связывания (в центре, синий/оранжевый/желтый) и ее потенциальными партнерами. Круги представляют собой уровень связывания сродства белковых комплексов. Рисунок иллюстрирует сложность общей проблемы «идентификации белковых партнеров» в рамках проекта Help Cure Muscular Dystrophy - этап 2, где потенциальные партнеры должны быть идентифицированы и отобраны в очень большом пространстве взаимодействий.
Дорогие волонтеры проекта Help Cure Muscular Dystrophy (Phase 2),
Я пишу, чтобы известить об анализе набора данных стыковочных конформациях, созданных проектом Cure Muscular Dicrophy (HCMD2). Целью проекта было исследование белково-белковых взаимодействий для более чем 2200 белков, чьи структуры известны, с особым упором на те белки, которые играют роль в нервно-мышечных заболеваниях.
В настоящее время анализируется полный набор кросс-стыковочных данных, содержащий эти белок-белковые взаимодействия. Для этого мы используем различные вычислительные инструменты, которые мы разработали параллельно с расчетами, выполненными в World Community Grid.
JET2 Viewer: репозиторий белковых интерфейсов
Мы создали базу данных с белковыми интерфейсами для научного сообщества. Эта база данных, называемая JET2, является важной вехой для идентификации белковых партнеров. Почему это важно? Анализ данных HCMD1 и HCMD2 подчеркивает, что точное описание интерфейсов белка имеет решающее значение для распознавания партнеров по белкам, а база данных дает очень точный набор таких прогнозов. Улучшение по-прежнему необходимо, но результаты уже очень точны.
В настоящее время мы концентрируемся на извлечении полезной информации о мышечной дистрофии из набора данных JET2, которую ваша поддержка помогла построить. Как вы знаете, прогресс в исследованиях идет медленно, но мы делаем определенные успехи и ожидаем, что методологический подход, используемый в HCMD2, будет очень успешным в крупномасштабной идентификации белковых партнеров при разрушении на уровне белка-остатка! (Белок-остаток представляет собой часть белка. *)
BIS2Analyzer: сервер, помогающий нашему анализу данных
Мы выпустили сервер под названием BIS2Analyzer, чтобы помочь идентифицировать сигналы взаимодействия на основе анализа коэволюции в белках или между белковыми партнерами. Этот сервер, который также открыто доступен для научного сообщества, важен, поскольку он может помочь идентифицировать сигналы взаимодействия с белком. Было полезно восстановить первую сеть взаимодействия белка с белком для вирусов (см. Ссылку ниже о восстановлении сети для вируса гепатита C).
Поблагодарив вас снова за ваши вклады. Я буду держать вас в курсе наших результатов. Будьте уверены, что мы добиваемся прогресса, благодаря вашей поддержке!
С наилучшими пожеланиями,
Алессандра
JET2Viewer:
H. Ripoche, E. Laine, N. Ceres, A. Carbone. JET2 Viewer: база данных прогнозируемых множественных, возможно перекрывающихся сайтов белка-белка для структур PDB, исследование нуклеиновых кислот (2016) doi: 10.1093/nar/gkw1053
E.Laine, A.Carbone. Локальная геометрия и эволюционное сохранение белковых поверхностей показывают множественные пятна распознавания в белок-белковых взаимодействиях, PLoS Computational Biology, 11 (12), e1004580, 2015.
BIS2Analyzer:
F. Oteri, F. Nadalin, R. Champeimont, A. Carbone, BIS2Analyzer: сервер для совместного эволюционного анализа консервативных семейств белков, Nucleic Acids Research, 2017.
R. Champeimont, E. Laine, S.-W. Hu, F. Penin, A. Carbone, Анализ коэволюции генома вируса гепатита C для идентификации структурной и функциональной сети зависимостей вирусных белков, Scientific Reports, Nature Publishing Group, 6: 26401, 2016.
* Примечание: «Уровень остатка» означает, что мы не только идентифицируем, является ли белок P партнером белка Q (ответ да/нет), но и в каком положении они взаимодействуют, определяя, какой остаток белка P может взаимодействовать с каким остатком белка Q.
новость на англ.
29 июня 2017 г.
Кратко
В этом письме к добровольцам д-р Алессандра Карбоун, главный исследователь проекта Help Cure Muscular Dystrophy ("помогите вылечить мышечную дистрофию") объявляет о выпуске двух новых инструментов в открытом доступе, которые могут ускорить работу исследователей, работающих над взаимодействием белков.

Картинка выше - рендеринг белковой структуры с тремя участками взаимодействия связывания (в центре, синий/оранжевый/желтый) и ее потенциальными партнерами. Круги представляют собой уровень связывания сродства белковых комплексов. Рисунок иллюстрирует сложность общей проблемы «идентификации белковых партнеров» в рамках проекта Help Cure Muscular Dystrophy - этап 2, где потенциальные партнеры должны быть идентифицированы и отобраны в очень большом пространстве взаимодействий.
Дорогие волонтеры проекта Help Cure Muscular Dystrophy (Phase 2),
Я пишу, чтобы известить об анализе набора данных стыковочных конформациях, созданных проектом Cure Muscular Dicrophy (HCMD2). Целью проекта было исследование белково-белковых взаимодействий для более чем 2200 белков, чьи структуры известны, с особым упором на те белки, которые играют роль в нервно-мышечных заболеваниях.
В настоящее время анализируется полный набор кросс-стыковочных данных, содержащий эти белок-белковые взаимодействия. Для этого мы используем различные вычислительные инструменты, которые мы разработали параллельно с расчетами, выполненными в World Community Grid.
JET2 Viewer: репозиторий белковых интерфейсов
Мы создали базу данных с белковыми интерфейсами для научного сообщества. Эта база данных, называемая JET2, является важной вехой для идентификации белковых партнеров. Почему это важно? Анализ данных HCMD1 и HCMD2 подчеркивает, что точное описание интерфейсов белка имеет решающее значение для распознавания партнеров по белкам, а база данных дает очень точный набор таких прогнозов. Улучшение по-прежнему необходимо, но результаты уже очень точны.
В настоящее время мы концентрируемся на извлечении полезной информации о мышечной дистрофии из набора данных JET2, которую ваша поддержка помогла построить. Как вы знаете, прогресс в исследованиях идет медленно, но мы делаем определенные успехи и ожидаем, что методологический подход, используемый в HCMD2, будет очень успешным в крупномасштабной идентификации белковых партнеров при разрушении на уровне белка-остатка! (Белок-остаток представляет собой часть белка. *)
BIS2Analyzer: сервер, помогающий нашему анализу данных
Мы выпустили сервер под названием BIS2Analyzer, чтобы помочь идентифицировать сигналы взаимодействия на основе анализа коэволюции в белках или между белковыми партнерами. Этот сервер, который также открыто доступен для научного сообщества, важен, поскольку он может помочь идентифицировать сигналы взаимодействия с белком. Было полезно восстановить первую сеть взаимодействия белка с белком для вирусов (см. Ссылку ниже о восстановлении сети для вируса гепатита C).
Поблагодарив вас снова за ваши вклады. Я буду держать вас в курсе наших результатов. Будьте уверены, что мы добиваемся прогресса, благодаря вашей поддержке!
С наилучшими пожеланиями,
Алессандра
JET2Viewer:
H. Ripoche, E. Laine, N. Ceres, A. Carbone. JET2 Viewer: база данных прогнозируемых множественных, возможно перекрывающихся сайтов белка-белка для структур PDB, исследование нуклеиновых кислот (2016) doi: 10.1093/nar/gkw1053
E.Laine, A.Carbone. Локальная геометрия и эволюционное сохранение белковых поверхностей показывают множественные пятна распознавания в белок-белковых взаимодействиях, PLoS Computational Biology, 11 (12), e1004580, 2015.
BIS2Analyzer:
F. Oteri, F. Nadalin, R. Champeimont, A. Carbone, BIS2Analyzer: сервер для совместного эволюционного анализа консервативных семейств белков, Nucleic Acids Research, 2017.
R. Champeimont, E. Laine, S.-W. Hu, F. Penin, A. Carbone, Анализ коэволюции генома вируса гепатита C для идентификации структурной и функциональной сети зависимостей вирусных белков, Scientific Reports, Nature Publishing Group, 6: 26401, 2016.
* Примечание: «Уровень остатка» означает, что мы не только идентифицируем, является ли белок P партнером белка Q (ответ да/нет), но и в каком положении они взаимодействуют, определяя, какой остаток белка P может взаимодействовать с каким остатком белка Q.
новость на англ.