Счастливого Рождества!
Dec. 26th, 2022 12:11 amУважаемые участники,
Желаем вам счастливого Рождества и счастливого Нового года!
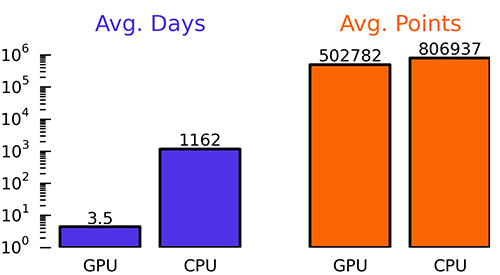
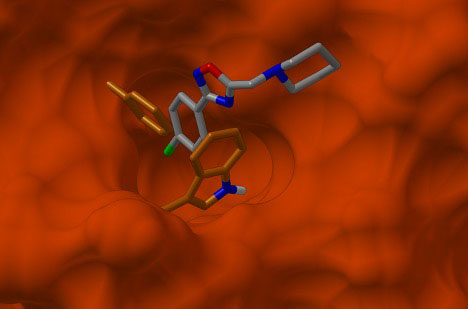
С вашей помощью мы выполнили большое количество компьютерного моделирования. Мы собираемся продолжать и верим, что результаты приведут к прогрессу в разработке лекарств! На данный момент у нас есть новый выпуск CmDock, и мы будем использовать его, начиная со следующей цели. Вот иллюстрация моделирования, выполненного в проекте, визуализированная с помощью PyMOL. Вы видите большую молекулу (это мишень, белок TMPRSS2) с молекулой во много раз меньшей. Малые молекулы являются лигандами, каждая из них может взаимодействовать с мишенью по-своему, со своей энергией связи.
Чем точнее лиганд связывается с мишенью, тем выше шанс, что он сможет изменить функционирование большой молекулы. Например, показанный на изображении фермент TMPRSS2 необходим для химической реакции, позволяющей коронавирусу проникнуть в клетки организма и, соответственно, вещество, способное подавить нормальную работу этого фермента, может затруднить или заблокировать распространение коронавируса. В ходе моделирования на ваших компьютерах тестируются различные лиганды из библиотеки, и лучшие из них отбираются для обработки на следующих этапах разработки лекарств.
Всего наилучшего,
Наталья, Марко, иней, Чртомир

на англ.
Желаем вам счастливого Рождества и счастливого Нового года!
С вашей помощью мы выполнили большое количество компьютерного моделирования. Мы собираемся продолжать и верим, что результаты приведут к прогрессу в разработке лекарств! На данный момент у нас есть новый выпуск CmDock, и мы будем использовать его, начиная со следующей цели. Вот иллюстрация моделирования, выполненного в проекте, визуализированная с помощью PyMOL. Вы видите большую молекулу (это мишень, белок TMPRSS2) с молекулой во много раз меньшей. Малые молекулы являются лигандами, каждая из них может взаимодействовать с мишенью по-своему, со своей энергией связи.
Чем точнее лиганд связывается с мишенью, тем выше шанс, что он сможет изменить функционирование большой молекулы. Например, показанный на изображении фермент TMPRSS2 необходим для химической реакции, позволяющей коронавирусу проникнуть в клетки организма и, соответственно, вещество, способное подавить нормальную работу этого фермента, может затруднить или заблокировать распространение коронавируса. В ходе моделирования на ваших компьютерах тестируются различные лиганды из библиотеки, и лучшие из них отбираются для обработки на следующих этапах разработки лекарств.
Всего наилучшего,
Наталья, Марко, иней, Чртомир

на англ.